▼もくじ
はじめに
弊社の次世代型発現解析マイクロアレイ「Clariom D」は、総プローブ数600万以上を誇る超高密度の多機能マイクロアレイであり、弊社の発現解析製品の中でまさにフラッグシップと呼ぶにふさわしいモデルとなっています。Clariom Dは従来の発現解析に加え、選択的スプライシング解析やlong non-coding RNA (lncRNA) 解析など、トランスクリプトームレベルでの解析にも威力を発揮します。ここではClariom Dの圧倒的な解析性能についてまとめてみました。
圧倒的なプローブ数で転写領域をカバー
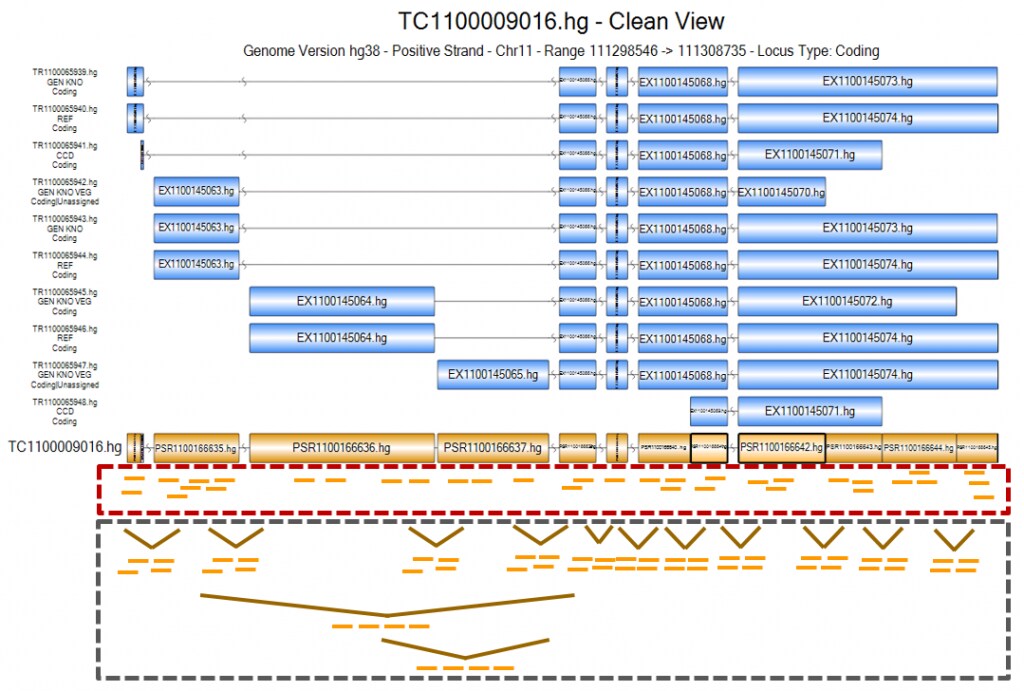
Clariom Dの性能を語る上で欠かすことのできないのが、そのプローブ数でしょう。600万を超えるプローブが遺伝子発現領域を広範囲にわたりカバーします。下図はClariom Dに搭載された各プローブが遺伝子のどの配列に対応しているかを示したものです。
上図はCOLCA2 (Colorectal Cancer Associated 2) 遺伝子のエクソン・イントロン構造とプローブの位置関係を模式的に示したものです。この図の赤い枠線で囲まれた部分がエクソン部分に対応しているプローブ群です。この図から分かるように、Clariom Dのプローブは遺伝子全体にわたってほぼ全てのエクソンに対応するように設計されています。また上図のグレーの線で囲まれた部分は、エクソンとエクソンの接合部、いわゆるエクソンジャンクション部に設計されたプローブです。このプローブにより、選択的スプライシングの解析精度が上がります。
このようにClariom Dでは一つの遺伝子に対して数十ものプローブが設計されているため、単なる遺伝子レベルの発現解析だけにとどまらない、トランスクリプトームレベルによる解析が可能となっているのです。
ちなみに、弊社以外のマイクロアレイでは多くの場合、1遺伝子をたった一つのプローブで解析しています。たった一つのプローブだけで1遺伝子を見ているということは、仮にそのプローブが何らかの理由で機能しなかった場合は、その遺伝子の情報が全く得られないことを意味しています。また1つのプローブデータでは統計的な処理ができませんので、データの信頼性に疑問が残ります。
このようなデメリットにもかかわらず単一プローブで解析せざるを得ないのは、マイクロアレイに搭載できるプローブ数に限界があるためです。一般的なマイクロアレイに搭載できるプローブ数は数万個程度しかないため、1遺伝子に複数のプローブを割り当ててしまうと解析できる遺伝子数が減ってしまうのです。
弊社のマイクロアレイは半導製造技術を応用しており、プローブ数が600万を超えるような超高密度マイクロアレイを作成できます。これにより、Clariom Dのように1遺伝子に数十ものプローブを設計してもなお数万にも及ぶ遺伝子の解析が可能なのです。
Clariom Dで解析可能な遺伝子
Clariom Dのヒト、マウス、ラットそれぞれの製品で解析できる遺伝子数などは下記の通りとなっています。
| Content summary | Human | Mouse | Rat |
| Genes※ | >134,700 | >66,100 | >68,900 |
| Transcripts※ | >542,500 | >214,900 | >495,200 |
| Exons※ | >948,300 | >498,500 | >320,400 |
| Exon-exon splice junctions※ | >484,900 | >282,500 | >293,700 |
| Total probes※ | >6,765,500 | >6,022,300 | >5,946,400 |
| Probes targeting exons※ | >4,781,200 | >4,895,600 | >4,780,700 |
| Probes targeting exon-exon splice junctions※ | >1,984,300 | >1,126,700 | >1,165,700 |
| Probe length(bases) | 25 | 25 | 25 |
| Probe feature size | 5μm | 5μm | 5μm |
| Background probes | Antigenomic Set | Antigenomic Set | Antigenomic Set |
※2016年4月時点での代表的なアノテーションの数です。10の位以下を切り捨てて表示しています。
例えばヒト用のClariom Dは134,700以上の遺伝子を解析でき、この中には542,500以上のスプライシングバリアントが含まれています。プローブの設計にあたっては15種類にも及ぶデータベースを利用しており、現在までに知られているスプライシングバリアントを網羅しています。

またコーディング遺伝子のみならず、ノンコーディング (non-coding) 遺伝子に関するプローブも豊富に含まれているため、バイオマーカー探索などのための情報を逃しません。
RNA-Seqを凌ぐ精度で低発現遺伝子もしっかり解析
近年、次世代シーケンサー(NGS)の性能が向上するにつれて遺伝子の発現解析をNGSでおこなう、いわゆるRNA-Seqのテクノロジーが広く用いられるようになってきました。発現量だけでなく配列情報も同時に得られるRNA-Seqは、まさに次世代のテクノロジーとして様々な場面で活用され始めています。一方でRNA-Seqを使いこなすためには、その特徴を予めしっかりと把握しておくことが肝心です。場合によっては思っていたような性能が出ずに、時間とコストを無駄にしてしまいかねません。
RNA-Seqの性能を端的に表したものとして、下図のようなデータがあります。

図5 RNA-Seqとマイクロアレイによる発現解析の比較。※Xu, W., et al. Human transcriptome array for high throughput clinical studies. Proc Natl Acad Sci USA 108(9):3707–3712 (2011).
上図はRNA-Seqとマイクロアレイによる発現解析を比較したものです。図の横軸は遺伝子の発現量を、縦軸は測定値のばらつきの大きさを表しています。
図の左上、1500万と赤字で書かれているグラフに注目してください。この赤字はRNA-Seqのリード数を示しています。マイクロアレイは低発現の遺伝子でも測定値のばらつきが小さいのに対し、RNA-Seqでは発現量が低くなるにつれて測定のばらつきがどんどん大きくなっていることが分かります。このように、リード数が少ない条件でのRNA-Seqでは低発現の遺伝子を精度よく解析できません。もしもRNA-Seqで低発現遺伝子を高精度で解析したければ、リード数を増やしていく必要があります。図の右下のように4.8億リードまでリード数を増やすことにより、ようやくマイクロアレイと同程度の解析性能を持つことが分かります。
一般的におこなわれているRNA-Seqではリード数にして4000万 (40 million) 程度で解析することが多いようです。上述したとおり、低発現の遺伝子まで精度よく測定するためには少なくともこの10倍以上のリード数が必要です。それはすなわち、一般的なRNA-Seqに比べて10倍のコストがかかることを意味しています。RNA-Seqでしか得られない情報が必要であればこのコストは必要経費として割り切れますが、もしもデータベース上にある既存遺伝子の解析が主な目的であるのならば、Clariom Dを用いた方がはるかに高精度かつ低コストで解析できるのです。
遺伝子レベルを超えた、トランスクリプトームレベル解析を実現
Clariom Dは従来のマイクロアレイで一般的におこなわれている遺伝子レベルの発現解析のほかに、選択的スプライシングによるスプライシングバリアント解析や、long non-coding RNA (lncRNA) 解析など、トランスクリプトームレベルでの解析をおこなうことができる次世代型マイクロアレイです。ここでは、Clariom Dによるトランスクリプトームレベル解析についてご紹介したいと思います。
選択的スプライシングによるスプライシングバリアント解析
Clariom Dの超高密度プローブは遺伝子領域をくまなくカバーするだけでなく、エクソンジャンクション部にも設計されています。これによりエクソンレベルでの精密な発現解析が可能となり、選択的スプライシングによるスプライシングバリアント解析をおこなうことができます。
例えば冒頭のCOLCA2遺伝子は、最新のデータベースの知見により10種類のスプライシングバリアントが存在していることが知られています。

Clariom Dは遺伝子の発現レベルに加え、各エクソンごとの発現レベルも同時に解析します。専用の解析ソフトを用いれば、サンプル中に含まれるバリアントのうち、どのタイプがもっとも多く発現しているかを予測できます。
long non-coding RNA (lncRNA) 解析
Clariom Dにはlong non-coding RNA (lncRNA) を解析するためのプローブが数多く搭載されています。下記の表はClariom Dを設計する際に用いられたデータベースの一覧と、採用された遺伝子数を表したものです。
| Data sources | Genes | ||
| Human | Mouse | Rat | |
| Ensembl | >57,500 | >57,500 | >25,300 |
| VEGA | >48,500 | >23,200 | – |
| NONCODE | >55,900 | >42,000 | >500 |
| lncRNAWiki | >50,000 | – | – |
| UCSC Genes | >43,800 | >26,300 | – |
| AceView | >41,100 | – | >30,900 |
| miTranscriptome | >34,500 | – | – |
| RefSeq | >25,600 | >23,500 | >16,900 |
| MGC | >17,200 | >17,400 | >6,400 |
| MGI | – | >25,800 | – |
| RGD | – | – | >30,300 |
| Consensus CDS | >18,500 | – | – |
| RNA Central | >17,200 | – | – |
| circBase | >12,200 | – | – |
| Human Body Map | >10,200 | – | – |
| lincRNAdb | >80 | >60 | >10 |
| Publication-specific gene sets | >3,000 | >10,000 | >10,290 |
| Non-overlapping orthologous mouse gene and transcript models | – | – | >21,500 |
この表から明らかなようにClariom Dにはnon-coding RNAのデータベースを元にして数多くのプローブが設計されています。これにより、今まで見過ごされてきたlncRNAの発現変動を確実にとらえることができます。
一般的にlncRNAの発現レベルはかなり低いため、先述したRNA-Seqで解析をするためには相当数のリード数を読む必要があり、いわゆるDeep Sequencingが必要と考えられています※。一方でDeep Sequencingは解析コストが増大するばかりでなく、データ解析のための手間や時間もかかってしまいます。Clariom Dは専用の解析ソフトによりどなたでも簡単にlncRNAの発現を解析できるため、lncRNAをターゲットとした研究に大きな威力を発揮します。
※Liu, Y., et al. Evaluating the impact of sequencing depth on transcriptome profiling in human adipose. PLoS One 8(6):e66883 (2013).
まとめ
今回は次世代型超高密度マイクロアレイClariom Dの特徴と性能についてみてきました。
現在、次世代シーケンサーを用いた発現解析は流行といっていいレベルで広範囲にわたって実施されていますが、RNA-Seqでできること、できないことを見極めた上で実験をしている人は意外と多くありません。限られた研究予算の中で真の意味での価値あるデータを出すためには、時流にとらわれない合理的な判断が求められています。Clariom Dは、未来に繋がるデータを求める研究者にとって最善の結果をもたらすことでしょう。
研究用にのみ使用できます。診断目的およびその手続き上での使用はできません。